Actualités
Interview Eric Rivals et Alexandre David
Publié le
Du développement d’outils à la conquête des marchés, une collaboration durable
Cartes d’identité :
|
|
|
|
|
|
|
- statut : Directeur de Recherche en biologie moléculaire à l’INSERM
- formation : Doctorat en immunologie, Post-doctorat sur le contrôle post-transcriptionnel de l’expression des gènes
- thématique de recherche : Étude des modifications chimiques des ARN dans les cellules souches cancéreuses
- poste : Chef de l’équipe Translation and Cancer à l’institut de Génomique Fonctionnelle (Montpellier)
|
Entretien
Comment a débuté votre collaboration ?
Il y a une dizaine d’années, à son retour en France, Alexandre a monté son groupe de recherche sur la régulation de l’expression des gènes au niveau traductionnel. C’était à l'époque un domaine émergent et le développement d’outils bioinformatiques était nécessaire. La prise de contact avec Eric, après qu’il ait assisté à un de ses séminaires sur l’analyse de transcriptome, a mené à des premiers travaux exploratoires sur la mise au point et l'analyse de Ribo-seq chez l'humain, avec la proposition d'une approche originale et d'un logiciel pour une question classique en biologie : le biais d'usage des codons.
Comment a évolué votre collaboration au fil du temps ?
Au fil des années, les projets plutôt théoriques ont laissé place à des projets ayant des objectifs plus appliqués en cancérologie, mais aussi à d’autres projets fondamentaux, notamment grâce à l’obtention d’un financement LabEx pluridisciplinaire (biologie, physique, maths et bioinformatique), multi-laboratoires pour un projet méthodologique qu’Eric a dirigé durant 4 ans. La complémentarité des expertises de chacun a permis d’explorer des questions complexes relatives à l’acquisition de résistance aux traitements dans les cancers colorectaux au travers de l'analyse conjointe du transcriptome et du traductome. Le but d’un tel projet est de découvrir les voies métaboliques dérégulées et les processus moléculaires à l'œuvre dans l’acquisition de la résistance.
Par la suite, ils ont élargi leur éventail de possibilités en cherchant des collaborateurs apportant de nouvelles compétences provenant d’autres disciplines. En effet, Christophe Hirtz, responsable de la Plateforme de Protéomique Clinique au CHU de Montpellier, a rejoint avec enthousiasme leurs projets. Ces derniers nécessitaient des connaissances en biochimie pour développer et appliquer des techniques de spectrométrie de masses, généralement utilisées en protéomique, à l’étude des modifications chimiques des nucléosides. En combinant l'analyse des modifications biochimiques des ARN isssus de prélévements tumoraux et des algorithmes d'apprentissage automatique, ils ont réussi à prédire le stade d'avancement du plus fréquent des cancers du cerveau, le gliome. Ces travaux ont donné lieu à un brevet et une publication. Afin d'améliorer la technique de détection, le projet a bénéficié des compétences d'une équipe de chimie aussi située à Montpellier. Cette collaboration multi-culturelle fonctionne ainsi depuis quelques années et émule les recherches dans chacune des disciplines impliquées.
Aujourd'hui, cet axe de recherche se développe dans un projet d'innovaton et de maturation technologique visant des applications cliniques pour le diagnostic précoce de cancers. Les objectifs différent de ceux de projets plus théoriques : il s'agit d'apporter des solutions technologiques d'aide à la décision aux médecins et cliniciens. Ils mènent des discussions préalables afin d'identifier les questions précises posées par les cliniciens, d'en comprendre les enjeux et voir comment cette nouvelle approche peut aider dans le traitement, la prise en charge, ou le suivi des patients.
Comment arrivez-vous à faire avancer vos projets communs ?
Le recrutement commun d’étudiant·e·s en thèse et en master est une étape essentielle à la faisabilité des projets. Ils recherchent en particulier des étudiant·e·s étant intéressé·e·s par acquérir la double compétence biologie/bioinformatique, qu’ils forment par la suite aux deux domaines. Ces doubles compétences sont d’ailleurs recherchées à l’interface avec les autres disciplines composant leur cluster de recherche (bioinformatique/biophysicien, biologie/chimie, etc.). Elles sont importantes à la fois pour les projets, car l’étudiant·e prend en compte les spécificités de chaque discipline et permet de faire un pont solide entre elles, mais aussi pour l’étudiant·e qui saura parler le langage de chaque discipline , ce qui n’est pas si courant, et facilitera les futures collaborations.
Leur message
Le mot d’ordre est d’être curieux, “Faut y aller !” , ne pas rester dans les sentiers battus. Une grande demande en bioinformatique est associée au séquençage haut-débit, mais il n’y a pas que ça comme technologie, la spectrométrie de masse par exemple a besoin de développement. “Regardez ce qui a été développé pour différentes technologies, ne restez pas dans une niche ! Il faut trouver la sienne !”, il faut sortir de sa zone de confort tous les 5-10 ans pour avoir un regard neuf, apprendre de nouvelles choses et pouvoir innover.
Il y a beaucoup de sujets passionnants et ça vaut le coût d’acquérir une vraie compréhension des deux domaines, ça demande des efforts mais qui sont rapidement récompensés. Les personnes ayant une bonne connaissance de l’informatique (algorithmique, apprentissage automatique, programmation) sont en particulier les bienvenues.
Ingénieure d’Études en Ingénierie Logicielle
Publié le
Le 19/06/2023
Carte d'identité:
|
|
- statut : Ingénieure d’Études en Ingénierie Logicielle
- formation : DEUG de Biologie, Licence de Biologie cellulaire et physiologie, Maitrise et Master 2 de bioinformatique de Paris 7
- poste : Ingénieure en développement bioinformatique, INRAE MaIAGE, Jouy-en-Josas
|
Entretien
Quels sont vos domaines de recherche ou d'expertise?
Pendant 9 ans, j’ai travaillé en tant qu’ingénieure en développement et déploiement d’applications sur la plateforme bioinformatique Migale de l’unité MaIAGE. Depuis 3 ans, j’ai rejoint l’équipe de recherche StatInfOmics au sein de la même unité. Je suis amenée à traiter des données assez hétérogènes allant des données « omiques » (génomique, transcriptomique, …) mais également des données issues de traitement automatique de la langue (text-mining) par exemple.
Concrètement, en quoi cela consiste ?
Mon travail consiste à développer des outils (bio)informatiques afin d’analyser ces données, les mettre à disposition de la communauté, les visualiser, …
Concrètement, je participe à la mise en place de workflow d’analyses. Le dernier en date est un workflow de détection de pseudogènes chez les bactéries. C’est une collaboration entre mon équipe mais également l’Institut Pasteur. Je mets également à disposition des bases de données, comme par exemple, Omnicrobe qui est une base de données dédiée aux habitats et phénotypes de micro-organismes. Sa particularité est qu’elle regroupe des données issues de différentes sources (PubMed, GenBank, DSMZ, CIRM). Je développe des interfaces web, comme Genoscapist qui permet de visualiser des données hétérogènes le long d’un génome. On peut en effet, parcourir des données d’annotation mais également des données d’expression.
En dehors de cette activité de développement qui occupe la majorité de mon temps, je suis également impliquée dans des actions de formation. En effet, depuis plus de 10 ans, je suis formatrice dans le cadre du cycle Bioinformatique par la pratique de la plateforme Migale.
Collaborez-vous avec des biologistes ? Quel type de collaboration est-ce ?
Une grande partie des outils que je développe sont à destination de microbiologistes. Je collabore donc étroitement avec eux afin de bien comprendre leurs questions de recherche et leurs besoins. La majorité des projets sur lesquels je travaille sont des collaborations entre bioinformaticiens, mathématiciens, informaticiens et microbiologistes. Chacun apporte ses compétences pour répondre à une problématique ciblée et partagée.
Quel avenir imaginez-vous pour la bioinformatique ?
La bioinformatique est un domaine qui a émergé il y a maintenant quelques années. C’est un domaine vaste, qui englobe plusieurs champs d’applications, que ce soit l’analyse de séquences, la bioinformatique structurale, l’analyse de réseaux, … C’est un domaine en constante évolution, avec l’émergence de nouveaux outils, de nouvelles méthodologies, … L’apport de la bioinformatique dans le domaine de la recherche n’est plus à démontrer. Outre les aspects de FAIRification des données et des analyses qui sont au cœur de la bioinformatique depuis quelques temps, il y a probablement des réflexions à avoir également côté RSE (Responsabilité Sociétale des Entreprises) dans notre domaine. Comment contribuer aux enjeux du développement durable dans le domaine de la bioinformatique ?
Votre message à destination des étudiants/jeunes bioinformaticiens ?
Ne vous fermez aucune porte, soyez curieuses et curieux ! Tout au long de votre parcours professionnel, que ce soit en stage ou même plus tard, explorez votre environnement. Visitez des laboratoires divers et variés : équipe de recherche, plateforme de service. Échangez avec des collègues de tout horizon : analyste, développeur, (bio)statisticien, mathématicien, biologiste, … Travaillez si possible sur des organismes différents : les plantes, les bactéries, l’Homme, … Et pour finir, n’oubliez pas, qu’en bioinformatique, on a la chance d’avoir une communauté très riche et plutôt bien structurée alors n’hésitez pas à la mobiliser si besoin et à partager.
Bilan des offres d’emplois et stages 2022
Publié le
Pour l’année 2022, , 1044 annonces ont été postées:
-
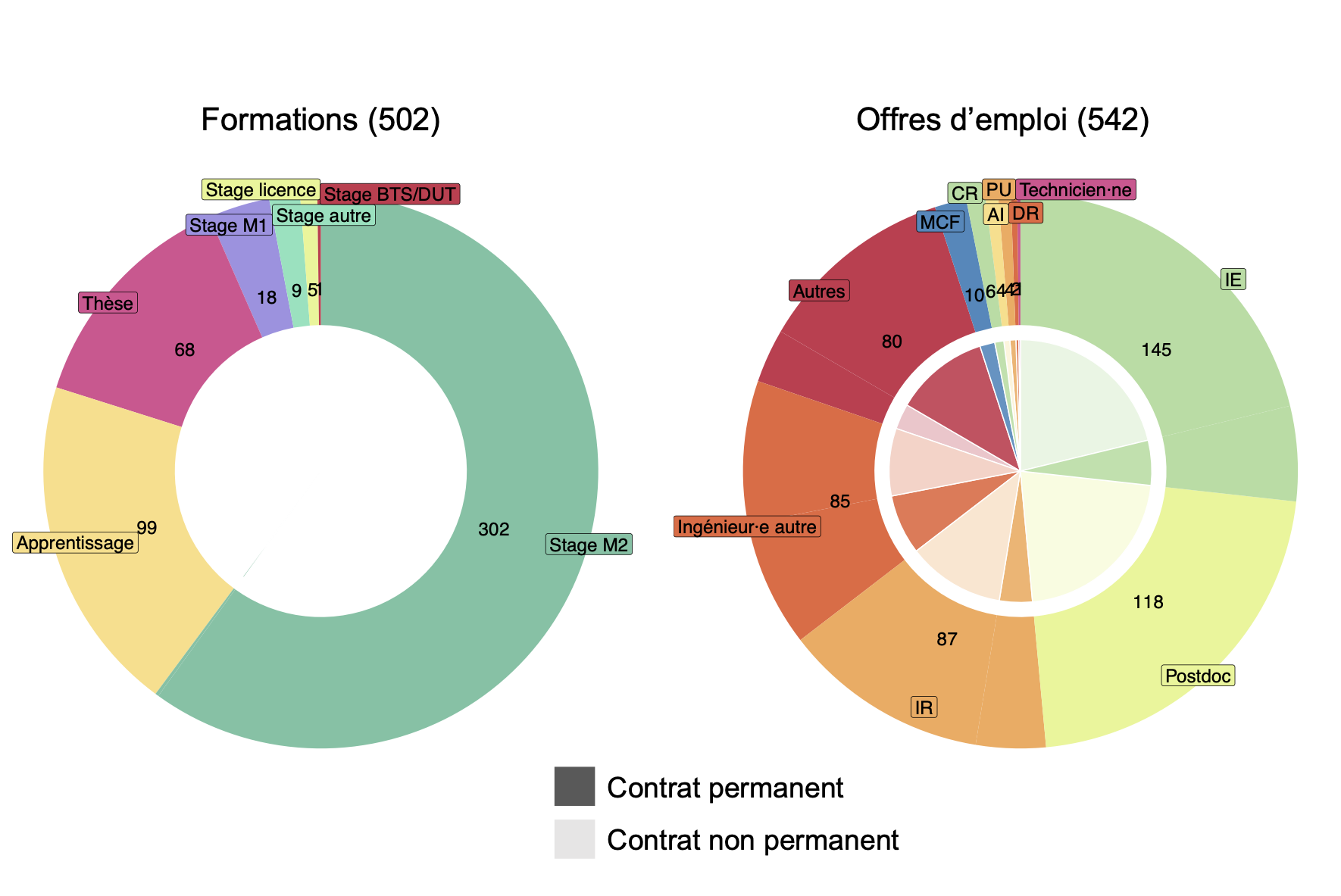
502 offres s’adressaient aux étudiants pour des stages de niveaux BTS à M2, les stages de M2 représentant la majorité des offres étudiantes (301), ainsi que 68 thèses, et 99 contrats d’apprentissage.
-
542 offres d’emploi dont 32.7% à durée indéterminée. Ces offres s’adressaient aux candidats en ingénierie ( de AI à IR en passant par les autres postes en ingénierie du monde du privé) pour 63% des offres en contrat court, et 52% en contrat à durée indéterminé, 32.6% et 11 % aux chercheurs et enseignant chercheur, et 5% et 36% aux autres postes.
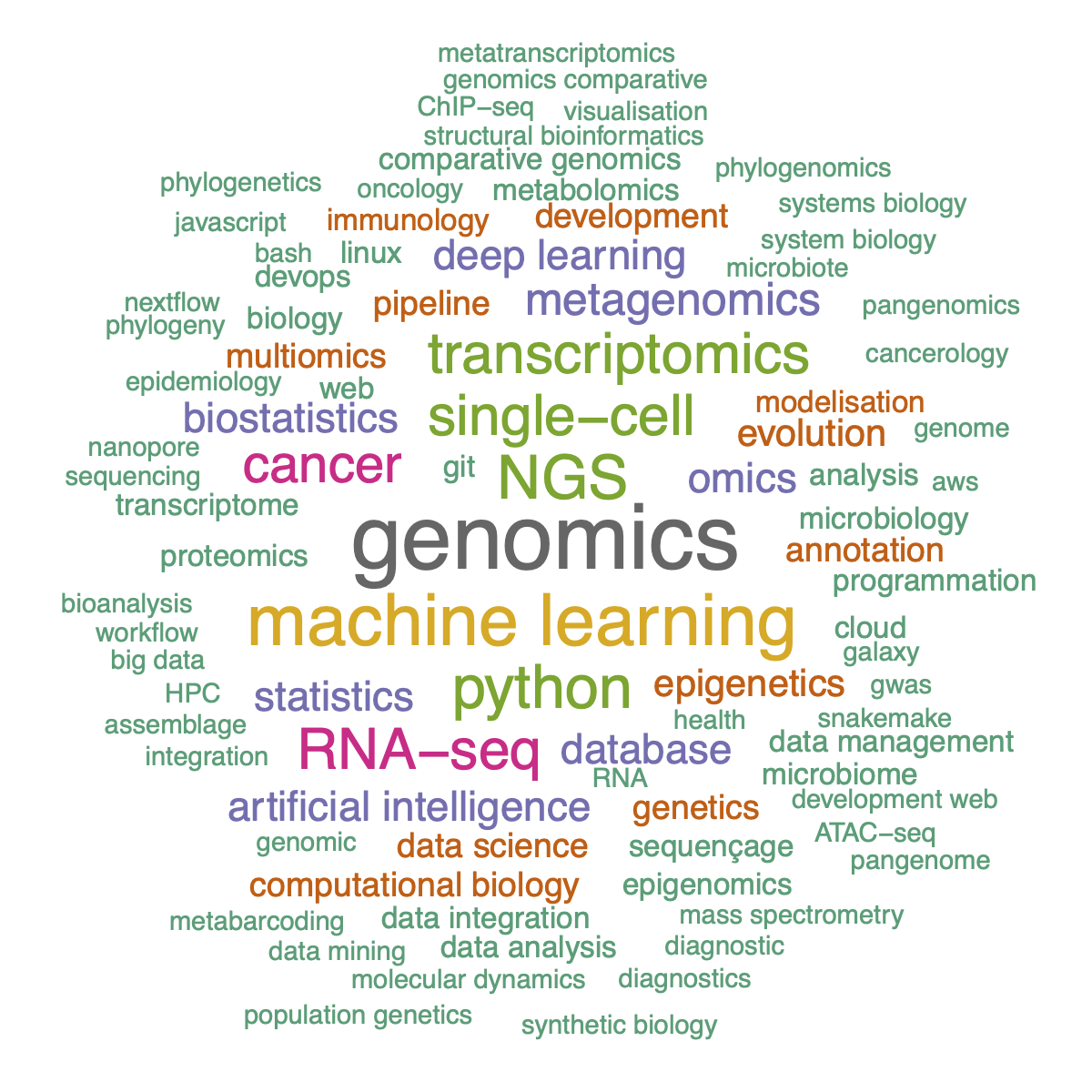
On retrouve majoritairement des offres du domaine de l’analyse de données séquences (méta-, épi-)génomiques et transcriptomiques et notamment via les technologies single-cell. Nous retrouvons également le développement de méthode dans les domaines du machine learning, de intelligence artificielle, du deep learning et de la modélisation (bio-)statistique.
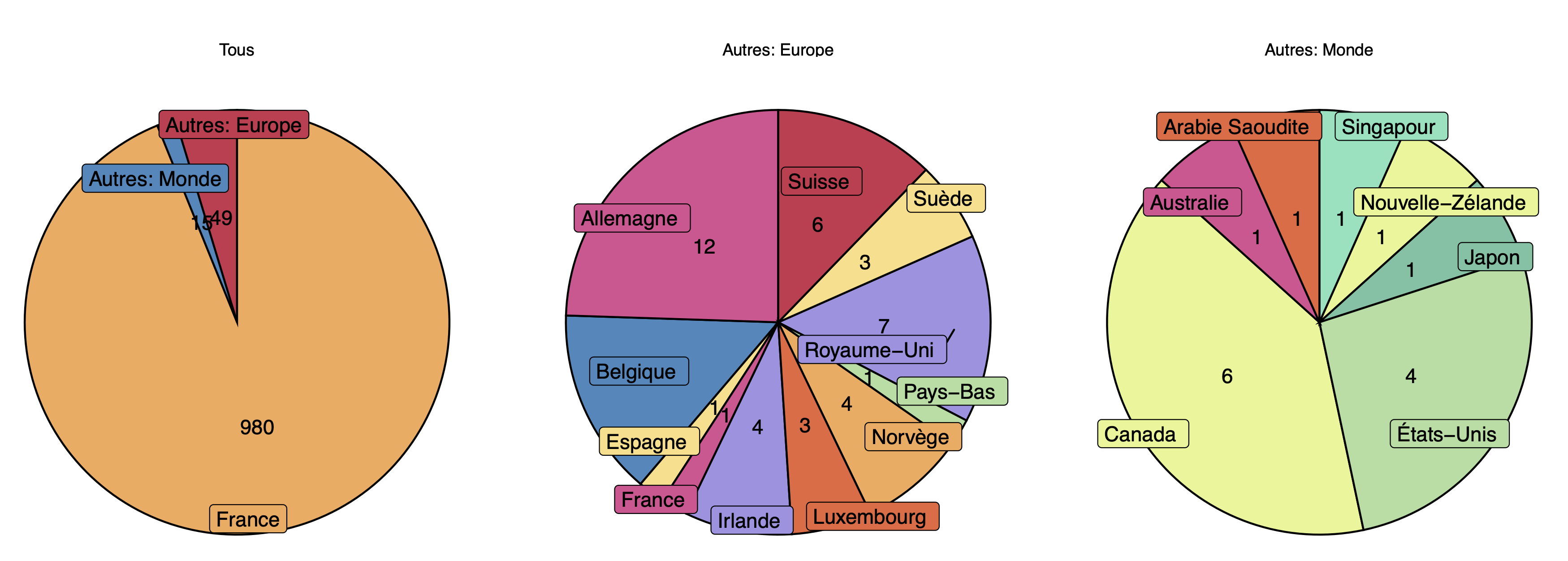
La majorité des annonces publiées concernent le territoire français (94% dont 2 en Guadeloupe), 49 de ces annonces concernent le territoire européen et 15 sont réparties ailleurs dans le monde.
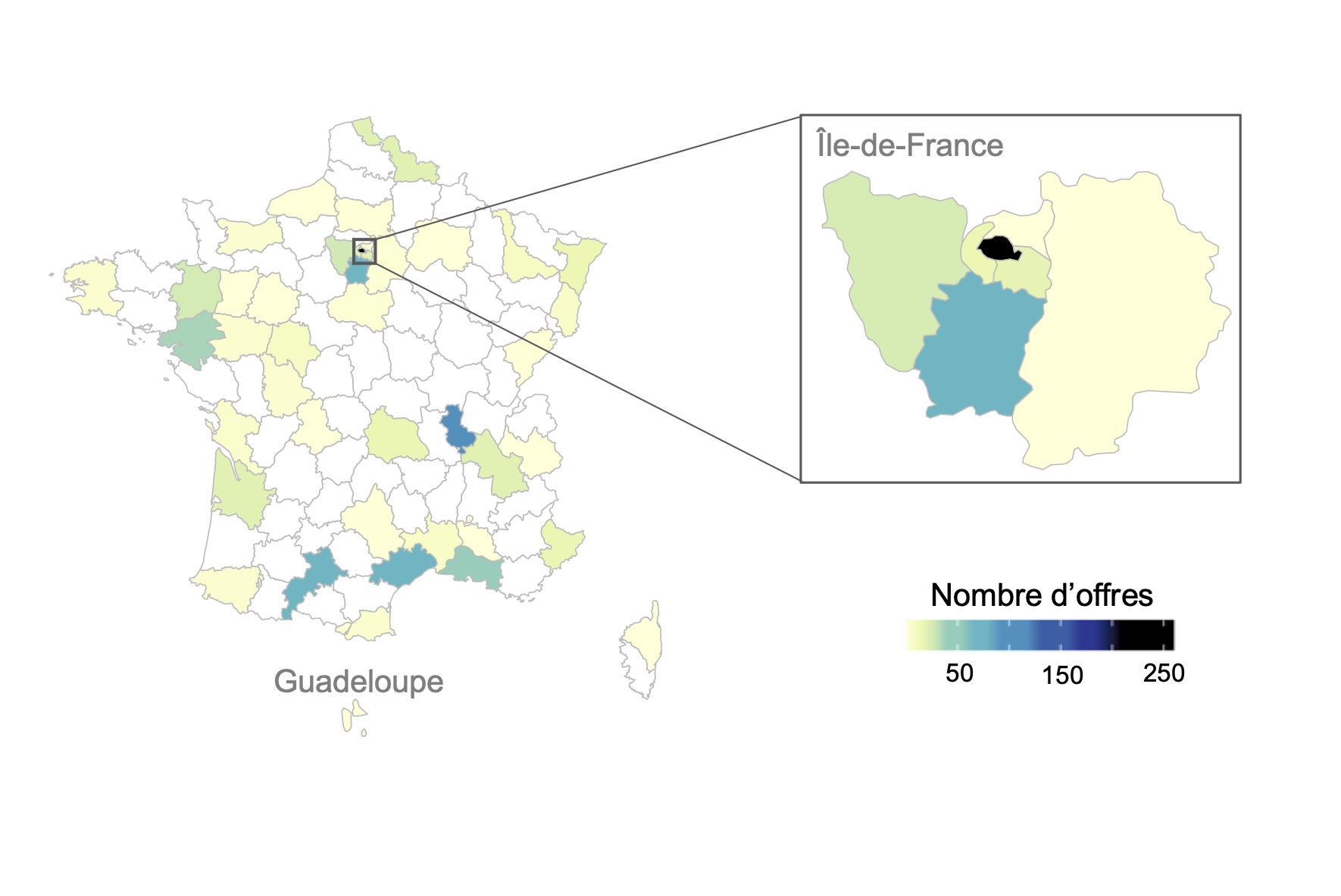
Plus précisément, en France, 42% des annonces concernent la région Île-de-France et 64% d'entre elles sont localisées à Paris. Environ 1/3 des offres se répartissent équitablement dans les régions Occitanie (17% dont 41% à Montpellier et 21% à Toulouse) et Auvergne-Rhône-Alpes (15% dont 63% à Lyon et 12% à Grenoble).
Pour 2023, à la date du 10 février, 124 offres ont déjà été mises en ligne sur le site de la SFBI.
Nous espérons que chaque offre a trouvé et trouvera un candidat. Bonne recherche à tous
Voeux 2023
Publié le
Bonjour à tous,
La SFBI vous souhaite une excellente nouvelle année 2023 et vous présente ses meilleurs vœux.
Quelques événements et actions vont marquer cette nouvelle année pour la SFBI. En effet ECCB/ISMB se déroulera à Lyon du 23 au 27 juillet, et la SFBI est partie prenante dans l’organisation de cette conférence durant laquelle nous aurons l’occasion de présenter la communauté bioinformatique française. Par ailleurs, nous ne renonçons pas à notre conférence annuelle : JOBIM se déroulera dans un nouveau format sur plusieurs sites (Montréal, Nancy, Nice, Pointe-à-Pitre, Roscoff, et Tours) du 27 au 30 juin.
Nous aimerions, pour cette année, débuter un répertoire des laboratoires dédiés à la bioinformatique ou accueillant des bioinformaticiens. Cette action a commencé par la mise en place d’un formulaire à remplir lors vos adhésions pour indiquer vos affiliations. Nous espérons ouvrir ce formulaire à tous les utilisateurs du site, pour in fine créer un répertoire et surtout une vue dynamique des structures d’accueil en France.
Enfin nous continuerons nos actions, en particulier sur la diffusion des offres d’emplois et stage, le soutien financier de déplacements ou d’organisations d’évènements, et la représentation de notre communauté au travers des différents réseaux en cours de structuration et association (COSSAF).
En espérant que la SFBI puisse continuer à être, à travers ses actions, une source de dynamisme pour notre communauté et de soutien pour chacun d'entre vous.
Anna-Sophie Fiston-Lavier, présidente
Matthias Zytnicki, vice-président
Maria Bernard, vice-présidente
Cyril Noël, trésorier
Yves Clément
Erwan Corre
Elodie Darbo
Sandra Dérozier
Delphine Potier
Bulletin SFBI 2022 #2
Publié le
La SFBI est heureuse de vous présenter son deuxième bulletin d'information 2022. Vous recevrez chaque trimestre un mail de synthèse sur les principales nouvelles de la communauté SFBI, ainsi que les activités de la SFBI et actions en cours. En complément de ce mail, nous vous invitons à découvrir et lire les actualités sur le nouveau site de la SFBI et celles du site des Bioinfomations. Ce dernier site regroupe les articles des actualités de la SFBI, les Jeunes Bioinformaticiens de France, Bioinfo-fr.net, IFB et du GDR-BIM.
Dans ce bulletin, vous trouverez des informations importantes sur JOBIM2023 et la conférence ISMB/ECCB2023.
-
JOBIM2022 :
-
Un grand merci à tous:
-
Plus de 500 participants, 42 présentations orales, 226 posters et 17 démos
-
Et aux organisateurs au top pour un retour au présentiel avec une superbe ambiance !
-
De très belles présentations des invités
-
Les actes et photos de l'évènement sont disponibles sur le site :
https://jobim2022.sciencesconf.org/resource/page/id/22
https://data-access.cesgo.org/index.php/s/LSZOb1SNn6ESMAG
-
Félicitations aux trois lauréats des prix SFBI :
-
Meilleure présentation : Brice Letcher, EMBL-EBI
-
Meilleur poster : Martina Rimoldi, EMBL-EBI
-
Meilleur poster/Demo : Oshma Chakoory, UCA
-
Et merci au Jury !
-
Félicitations aux 28 boursiers de la SFBI depuis le début de l'année 2022 ( dont 20 pour JOBIM2022 )
-
La SFBI est heureuse d'avoir pu soutenir deux familles et ainsi aider les parents bioinfo à participer à JOBIM
-
Deux conférences de Bioinformatique en France en 2023
ISMB/ECCB 2023 - 23/07/2023 au 27/07/2023
Tous les 4 ans, la Société Internationale de Bioinformatique (ISCB pour International Society of Computational Biology) se joint à la Communauté Européenne de Bioinformatique pour l’organisation d’une conférence en Europe. La conférence internationale ISMB-ECCB (Intelligent Systems for Molecular Biology - European conference on Computational Biology) 2023 se déroulera en France, à Lyon, du dimanche 23 au jeudi 27 juillet 2023. La tenue de cette conférence prestigieuse en France représente une occasion unique pour toute la communauté nationale de participer à une conférence internationale en Bioinformatique. Pour les jeunes bioinformaticiens, la conférence ISMB-ECCB 2023 est l’occasion de rencontrer la communauté internationale, et ainsi parfaire leur réseau, explorer d’autres thématiques de la bioinformatique et cela à moindre frais.
Malgré cette économie des frais de transport, les coûts d’inscriptions (même subventionnés) des conférences ISMB restent plus élevés que ceux des JOBIM. Afin de faciliter l'accès à cette conférence, la SFBI proposera des inscriptions à des tarifs préférentiels pour ses membres (pensez donc à adhérer à l’association). De son côté, en complément, le GDR-BIM proposera des bourses pour prendre en charge les frais de mission des doctorants et post-doctorants.
Contactée par l’ISCB qui pilote les conférences ISMB-ECCB, la SFBI a négocié avec les organisateurs de cet événement l’organisation d’une session “Bioinformatique Française”. Cette session planifiée durant la conférence ISMB-ECCB 2023 aura pour but de mettre en avant les spécificités de la bioinformatique en France avec des orateurs invités de notre communauté et un événement social local. Cette session sera pilotée par la SFBI avec le soutien d’autres structures de la bioinformatique française (GDR-BIM et IFB).
Pour cela, la présidente de la SFBI (Anna-Sophie Fiston-Lavier) a été invitée à siéger au comité de pilotage.
Pour plus d’informations, nous vous invitons à visiter le site : https://www.iscb.org/ismbeccb2023/
-
JOBIM2023 - 27/06/2023 au 30/06/2023
Par ailleurs après plusieurs discussions avec le collège d’experts et au sein de la SFBI, il a été décidé par le bureau de la SFBI de maintenir la conférence JOBIM en 2023. Elle aura lieu en amont du congrès ISMB-ECCB, du mardi 27 au vendredi 30 juin 2023, afin de permettre au plus grand nombre de partager leur travail avec la communauté bioinformatique; et cela toujours dans une atmosphère bienveillante et donc propice aux présentations des jeunes bioinformaticiens.
L’organisation des JOBIM requiert habituellement deux ans de préparation. Depuis quelques années, plusieurs villes de France candidatent auprès de la SFBI pour l'organisation des JOBIM en dehors des grandes villes habituelles. Dans ces conditions tardives, la SFBI a décidé de solliciter plusieurs de ces villes pour proposer en 2023 un congrès multisites. Ce JOBIM pilotée par la SFBI se tiendra en présentiel dans 6 lieux représentant des grandes régions avec des bioinformaticiens francophones: dans le Grand-Ouest (co-organisation Plouzané/Roscoff); dans le centre (co-organisation Tours/Orléans/Poitiers); dans le Nord-Est (co-organisation Nancy/Dijon); dans le Sud (organisation Nice); dans les Antilles Françaises (organisation Point-à-Pitre) et au Canada (organisation Montréal). Les organisateurs locaux s’efforceront de mettre en place des événements sociaux et des soirées de gala aux couleurs de la région. Les keynotes et orateurs sélectionnés pourront être dispatchés sur les différents sites permettant ainsi de conserver une bonne interaction (un contact humain). La SFBI encourage donc tous les participants à suivre la conférence en présentiel en rejoignant un des sites. Cette session se tiendra comme d’habitude sur 4 jours avec en fin de matinée les sessions posters et les conférences l’après-midi afin de prendre en compte le décalage horaire.
Les membres de la SFBI avec l’implication des locaux des différents sites investis dans cette nouvelle mission se sont répartis les tâches de la manière suivante :
-
Présidents de Comité d’Organisation : Erwan Corre, Emmanuelle Morin et Cyril Noël.
-
Présidents de Comité de Programme : Yves Clément, Delphine Potier et Matthias Zytnicki.
Sites - Contacts locaux
-
Grand Ouest - Erwan Corre (Roscoff), Cyril Noël (Plouzané)
-
Grand Est - Emmanuelle Morin (Nancy), Jonathan Kreplak (Dijon)
-
Sud - Karine Robbe-Sermesant (Nice)
-
Centre - Gaëlle Lefort (Tours), Bouziane Moumen (Potiers), Odile Rogier (Orléans)
-
Antilles Françaises - David Couvin (Guadeloupe - Pointe-à-Pitre)
-
Canada - Guillaume Bourque, Diallo Abdoulaye (Montréal)
Pour plus d’informations bientôt sur :
https://jobim2023.sciencesconf.org/