Actualités
Rencontre des réseaux ouest-africains et français autour de la bioinformatique
Publié le
Du 15 au 17 décembre 2025, une délégation de l’Institut Français de Bioinformatique (IFB/ELIXIR-FR), de la Société Française de Bioinformatique (SFBI) et de l’Université de Montpellier a été invitée au lancement du Réseau ouest Africain des Bioinformaticiens et Administrateurs de Systèmes (RABIAS), organisé par le centre de recherche d’excellence WAVE (Central and West African Virus Epidemiology for Food Security) et l’IRD (Institut de Recherche pour le Développement).

L’initiative RABIAS fait suite à la deuxième édition du Certificat International de Bioinformatique et Génomique (CIBiG) en Afrique de l’Ouest et à une formation au cluster HPC d’un groupe d’administrateurs systèmes de pays de la sous-région. Le réseau RABIAS a pour objectif de fédérer les communautés de bioinformaticiens, d’administrateurs systèmes et d’usagers de la région ouest africaine afin d’impulser la mutualisation de ressources humaines et matérielles et de stimuler la montée en compétence des équipes de recherche locales.
La rencontre réunissait à Abidjan (Côte d’Ivoire) les acteurs clés du séquençage, de la bioinformatique, et de l’administration systèmes des ressources de calcul de la région ouest africaine (Côte d’Ivoire, Burkina Faso, Bénin, Nigéria, Togo, Tchad, République Démocratique du Congo, Ghana, Guinée et Niger), ainsi que des représentants de l’African Society for Bioinformatics and Computational Biology (ASBCB) et du Pan African Bioinformatics Network (H3ABioNet).
Les trois journées alternaient présentations, retours d’expériences et ateliers en petits groupes afin de définir les besoins, les moyens et les priorités stratégiques. Les présentations scientifiques fournissaient un aperçu de travaux de recherche dans les domaines phares tels que génomique humaine, surveillance génomique des pathogènes, phytopathologie, résistances aux antimicrobiens, ainsi que les enjeux liés au déploiement d’infrastructures HPC distribuées.
Retours d’expériences des instances françaises invitées
Afin d’illustrer la complexité des problématiques liées au montage de réseaux, les acteurs invités ont partagé leurs expériences concernant la formation, l’infrastructure et la mutualisation des ressources (matériel et humains). L’IFB a présenté sa structuration en tant que fédération de plateformes régionales et que noeud français du réseau ELIXIR, l’organisation de son réseau national de ressources numériques (NNCR), et ses actions de formation continue et de développement des compétences. L’Université de Montpellier (UM) a présenté un panorama des formations diplômantes en France, suivi d’un focus sur son Master en Bioinformatique, mettant en avant l’apport des nouvelles approches pédagogiques des Montpellier Omics Days et du Bioinformatics Learning Lab. La SFBI a présenté son organisation et ses principales missions, en mettant l’accent sur ses interactions avec les autres acteurs de la bioinformatique française ainsi que d’autres réseaux internationaux de bioinformatique.

L’ensemble des discussions et des ateliers de travail ont permis d’identifier des priorités et des actions à mettre en œuvre pour le développement et la maturation du réseau RABIAS. À l’issue de cette rencontre un comité directeur inter-instituts et international a été identifié et des groupes de travail constitués par des participants ont pris l’engagement de mettre en œuvre les actions prioritaires planifiées pour 2026. Cette rencontre a permis de poser les premiers jalons du réseau et de montrer le dynamisme et l’implication de la communauté dans cette initiative. Rendez-vous est pris pour une future édition !

Boutique Tunetoo inacessible
Publié le
Nous vous informons que la boutique Tunetoo SFBI a été désactivée pour une durée indéterminée.
Le bureau de la SFBI essaie d'entrer en contact avec la société Tunetoo depuis maintenant plusieurs mois mais sans succés.
Tant que nous n'aurons pas réussi à avoir des informations de la part concernant notre boutique, celle-ci sera désactivée.
Nous nous excusons pour la gène occasionnée.
Maîtresse de conférence multi-facettes
Publié le
Le 18/12/2024
Carte d'identité:
|
Thomas-Chollier Morgane
|
- Statut : Maîtresse de conférence en délégation au CNRS à temps partiel
- Formation : Master1 en Biologie Cellulaire et Physiologie (Orléans) ; Master2 en Bioinformatique et Génomique (Strasbourg) ; Doctorat en Bioinformatique (Bruxelles) ; Post-doctorat en Bioinformatique (Berlin)
- Poste :
- Maîtresse de conférence à l’Ecole Normale Supérieure ; Responsable scientifique de la plateforme GenomiqueENS ; directrice adjointe de l’Institut Français de Bioinformatique (IFB) chargée du réseau des plateformes
|
Entretien
Quels sont vos domaines de recherche ou d’expertise ?
En tant que maîtresse de conférence, il y a deux volets à mon métier. Côté enseignement, j’ai une expertise pédagogique, à la fois en formation initiale pour les étudiant·es de mon établissement, mais aussi en formation continue. Je reste très motivée par le challenge d’enseigner la bioinformatique à un public issu de la biologie. Côté recherche, c’est en génomique ! Initialement en épigénomique, puis en transcriptomique. En rejoignant la plateforme GenomiqueENS, je me suis rapprochée des séquenceurs, et je reste encore aujourd’hui fascinée par ces machines et leur technologie.
En tant que responsable scientifique de cette plateforme, et comme une des directrices adjointes de l’Institut Français de Bioinformatique (IFB), mon expertise se situe autour du management, du pilotage stratégique, d’animation de réseau, et de représentation. Des activités dont je ne connaissais pas vraiment l’existence il y a encore quelques années !
Quel rôles exercez-vous ? Concrètement, en quoi cela consiste ?
Auprès des étudiant·es de l’ENS et des thésard·es que j’encadre, mon rôle est de les accompagner dans leurs apprentissages de la bioinformatique, en les suivant sur plusieurs années vers leur projet professionnel.
Au sein de la plateforme, mon rôle est d’assurer que l’équipe a tout ce qu’il faut pour mener leurs propres missions. J’apporte mon expertise scientifique pour nos projets de recherche et développement, basés sur les domaines d’expertises de nos ingénieur·es, en particulier la transcriptomique en lecture longue Nanopore.
Au sein de l’IFB, mon rôle est entre autres d’animer le réseau des plateformes, distribué sur toute la France. Concrètement, j’organise des réunions, et beaucoup de visioconférences !
Collaborez-vous avec des biologistes ? Quel type de collaboration est-ce ?
La plateforme permet aux chercheurs de réaliser leurs projets de transcriptomique, de l’échantillon jusqu’à l’analyse bioinformatique et la publication des résultats. Notre équipe comporte donc des ingénieur·es biologistes en charge de la partie expérimentale, et des ingénieur·es en bioinformatique. Collaborer avec des biologistes est donc au cœur même de notre organisation, et de nos activités avec les chercheurs.
Quel avenir imaginez-vous pour la bioinformatique ?
La bioinformatique n’échappe pas à la révolution qu’apporte l’intelligence artificielle. Dans les années à venir, le développement de nouvelles méthodes va être foisonnant ! Pour autant, l’accessibilité des outils bioinformatiques de pointe ne peut pas se restreindre aux seuls bioinformaticien·nes. Un des enjeux est d’assurer des interfaces adaptées aux biologistes, sur des infrastructures de calcul haute-performance. L’IFB joue un rôle clef en France sur ces aspects, et aussi pour former les biologistes à la bioinformatique, leur permettant ainsi de mieux communiquer avec les bioinformaticien·nes et de répondre à leurs questions biologiques. Enfin, un des enjeux de la bioinformatique est d’assurer la reproductibilité des analyses, la mise à disposition de données (vraiment) réutilisables, dans un contexte de science ouverte.
Votre message à destination des étudiants/jeunes bioinformaticien·nes ?
La communauté française en bioinformatique est vraiment dynamique et sympa. Ne restez pas seul·e dans votre coin ! Etape 1 : adhérez gratuitement à l’association Jebif, ils sont là pour vous donner plein de conseils et vous faire rencontrer d’autres jeunes bioinformacien·nes. Etape 2 : lisez le blog bioinfo-fr.net et découvrez leur canal discord. Etape 3 : élargissez encore un peu vos horizons en lisant la newsletter Les Bioinformations. Etape 4 : vous êtes en stage/thèse/contrat => rendez-vous à JOBIM, l'événement annuel de la bioinformatique piloté par la SFBI, pour y présenter vos travaux pour la première fois, et rencontrer la communauté. Etape 5 : envie de travailler avec d’autres bioinformaticien·nes ? Identifier les réseaux près de chez vous et les groupes de travail thématiques via le réseau métier MERIT.
Interview Anne Goelzer et Nadia Bessoltane
Publié le
Bioinformatique et Mathématiques: outils complémentaires pour explorer la biologie des systèmes
Cartes d’identité
|
|
|
|
- statut : ingénieure de recherche en biologie des systèmes au sein de l’unité INRAE MaIAGE en détachement à MIAT Toulouse.
- formation : diplôme d'ingénieur en Génie physique et systèmes embarqués spécialité automatique à l'ENSICAEN, DEA en Électronique Système Capteur Image à l'université de Caen, Doctorat en "Automatique/Informatique" de l'École Centrale de Lyon, Habilitation à diriger des recherches en Biologie des systèmes.
- poste : modélisation multi-échelle, analyse et simulation de systèmes biologiques (principalement bactéries, plantes).
|
|
- statut : ingénieure d’étude en bioinformatique au sein de l’Institut Jean-Pierre Bourgin (IJPB) à INRAE Versailles.
- formation : Licence en biologie cellulaire avec une option bioinformatique, Master "Cancérologie : Pharmacologie et thérapeutique" à l'Université Paris-Sud, Master "BioInformatique et BioStatistique" à l'Université Paris-Sud.
- poste : dans une équipe bioinformatique proposant des services allant de l’analyse de séquences génomiques (variants) en passant par l’analyse de données “omiques”, … sur les plantes et leurs pathogènes.
|
Entretien
Comment a débuté votre collaboration ?
A INRAE mais aussi dans d’autres instituts, il existe des structures appelées CATI pour Centre Automatisé de Traitement de l’Information. Ce sont des lieux de production informatique ou de services en soutien à une communauté scientifique ciblée. En 2019, Anne a participé au montage du CATI SysMics (Systems Biology for Omics) autour des approches de biologie des systèmes pour l’analyse intégrative de données multi-”omiques”. Nadia a rejoint ce dernier afin de travailler sur le déploiement de méthodes statistiques exploitant des données “omiques” hétérogènes à l’aide des processus biologiques. C’est dans ce cadre que les premiers échanges entre Anne et Nadia ont eu lieu. Les premiers travaux conjoints et plus concrets ont émergé en 2021. Cette année (2024), Nadia a décidé de débuter une thèse et Anne fera partie de ses encadrants.
Quelle est la question scientifique vous ayant mené à collaborer ensemble ?
Anne et quelques équipes de l’IJPB s'intéressent à la modélisation multi-échelle des plantes intégrant des informations au niveau cellulaire, multi-cellulaire, tissulaire dans le but de mieux prédire les phénotypes de plantes en fonction des stades d'évolution ou des conditions environnementales (conditions normales qui vont servir de contrôle et conditions environnementales contraintes telles que des stress hydriques, des stress azotés et la combinaison des deux). Un des aspects de cette recherche consiste à analyser les données “omiques” générées dans ce cadre, dans le but de les intégrer entre elles et avec les informations fonctionnelles. C'est dans ce cadre que Nadia intervient avec ses collègues : Delphine Charif, bioinformaticienne et Audrey Hulot, statisticienne.
Quels sont vos domaines de recherche / d'expertise ?
Anne travaille sur la modélisation de cellules, en particulier leurs réseaux métaboliques par des approches déterministes. Elle travaille également sur l’intégration de données à travers les modèles (estimation de paramètres des modèles par exemple). Anne a initialement travaillé sur les bactéries et transite vers les plantes depuis quelques années. Elle modélise des populations de cellules de différents tissus de plantes afin de les interconnecter pour aller jusqu’à la modélisation de la plante entière.
Nadia est spécialiste dans l'analyse de données issues de séquençage haut-débit, notamment RNA-seq, DNA-seq ainsi que les données protéomique et métabolomique. Elle effectue ces analyses dans le but d'extraire et de combiner un maximum d'informations qu'elle enrichit par la suite grâce aux nombreuses bases de connaissances biologiques.
Quel type de travaux effectuez-vous dans le cadre de votre collaboration ?
Tout d'abord, pour situer le contexte, cette question scientifique a été abordée dans le cadre d'un premier projet exploratoire financé par l’INRAE, puis est maintenant approfondie dans un projet ANR multidisciplinaire regroupant des experts en biologie (IJPB, IPSiM), mathématiques (MaIAGE, MICS) et bioinformatique (IJPB, MaIAGE). Les objectifs de ce projet consistent à mieux comprendre la stratégie d’allocation de ressources de la plante Arabidopsis thaliana en combinant modélisation mathématique multi-échelle et expériences biologiques. En particulier, il s’agit de construire des modèles quantitatifs de plantes entières, de les valider expérimentalement, mais aussi de développer les outils logiciels et les algorithmes nécessaires à leur génération, calibration et simulation. Ce projet implique donc des échanges fréquents entre Anne et les expérimentalistes afin de définir le plan expérimental adéquat pour remplir les objectifs scientifiques et acquérir les données expérimentales pour la calibration et la validation du modèle.
Dans ce cadre, Anne construit, calibre et valide des modèles quantitatifs intégrant une reconstruction détaillée des processus cellulaires à partir de l’analyse de la littérature et de discussions avec des experts de l'organisme "modèle”. Les modèles représentent le fonctionnement de l'organisme et permettent ainsi la prédiction de leur phénotype (flux, concentrations de protéines, vitesse de croissance) dans une condition expérimentale donnée, et sont spécialisés par type de tissus (c'est-à-dire spécifier les processus par type de tissus ou par type de population de cellules en extrayant de l’information dans des bases de données par exemple, afin de trouver des gènes qui vont coder pour certains types de réactions).
Nadia est impliquée à plusieurs niveaux du projet tout d'abord en mettant en place les pipelines pour spécialiser les modèles en utilisant les données “omiques”. Elle travaille également à l'alignement des informations extraites sur des graphes de connaissances permettant de faire ressortir des traits pertinents non identifiés par l'utilisation des méthodes statistiques "classiques". Aussi, l'analyse et l'intégration de données multi-”omiques” (RNA-seq, métabolomique, protéomique) vont permettre une interprétation plus approfondie pour des conditions expérimentales données.
Votre environnement de travail est-il majoritairement formé de bioinformaticien·ne·s, de biologistes ou est-ce en proportion équivalente ? Est-ce une équipe plateforme, une équipe de recherche ou autre ?
Nadia travaille au sein d’une équipe proposant des services en bioinformatique. Elle travaille étroitement avec une collègue bioinformaticienne ayant le même profil qu’elle, Delphine Charif et une autre collègue statisticienne, Audrey Hulot. Elles interagissent beaucoup avec les autres équipes de recherche de l’Institut afin de leur apporter leur aide sur le volet bioinformatique dans le cadre de leurs projets de recherche.
Anne est quant à elle rattachée à une équipe de recherche, en région parisienne, spécialisée en biologie des systèmes travaillant sur des approches déterministes. Cette équipe est principalement composée de mathématiciens. Anne est en détachement au sein d’une équipe de recherche toulousaine depuis plusieurs mois, qui est composée d’informaticiens, de bioinformaticiens et de statisticiens travaillant sur des méthodes statistiques en lien avec l’Intelligence Artificielle.
Quelle est la clé pour des échanges facilités ?
Au début de cette collaboration, les échanges n’étaient pas si évidents car chaque collaborateur utilisait un vocabulaire différent, propre à son domaine de compétences. Par exemple, le terme “modèle” n’a pas le même sens pour un modélisateur, un statisticien et un bioinformaticien. Selon Anne, qui a l’habitude de travailler à l’interface de diverses disciplines depuis plusieurs années, la clé est de clarifier le vocabulaire assez tôt lors des échanges. De plus, il lui semble essentiel de s’intéresser aux questions de l’autre discipline, de rendre intelligibles ses propres questions d'intérêt aux non-spécialistes (les questions de biologistes doivent être compréhensibles -et intéressantes- pour les matheux, et inversement), et par conséquent d’être prêt à investir du temps afin de bien comprendre les avantages et limites des approches de chacun. L’idée est d’être capable de faire progresser simultanément les questions des deux disciplines, et d’éviter qu’une discipline ne soit qu’au service de l’autre.
Leurs messages
Informez-vous bien sur les divers cursus en bioinformatique proposés par les différentes structures d’enseignement. Faites bien la différence entre un diplôme en biologie avec des notions abordées en bioinformatique et un diplôme complet en bioinformatique, les notions abordées ne seront pas autant approfondies dans les deux cas. Montrez également un intérêt pour le développement de méthodes et les nouveautés !
En effet, soyez curieux et restez ouverts. Soyez sensibles à d’autres disciplines que la vôtre. N’ayez pas peur des mathématiques ! Un peu de culture en mathématiques vous permettra de mieux appréhender certains challenges et de favoriser les échanges avec vos collaborateurs dans certains projets.
Boutique de goodies de la SFBI
Publié le
, dernière mise à jour le
Nous avons le plaisir de vous annoncer l’ouverture de la boutique officielle de goodies SFBI (https://sfbi.tunetoo.com/).
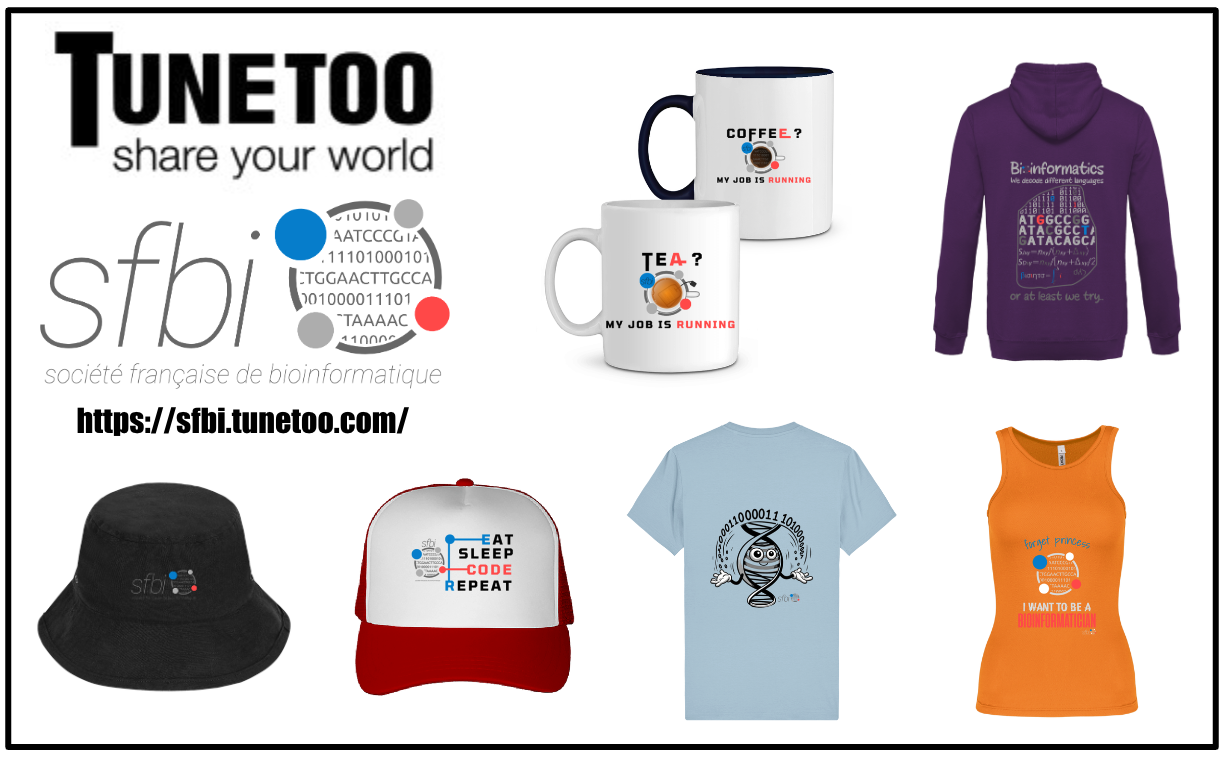
Vous y trouverez des tee-shirts, des sweat-shirts, des casquettes, des mugs, … avec les nouveaux visuels SFBI mais également les “anciens”. De nombreux modèles sont disponibles (avec le visuel sur le devant ou dans le dos) ainsi que différentes couleurs. Nous espérons que vous trouverez votre bonheur.
Nous en profitons pour remercier l’ensemble des participants au concours de visuels et féliciter une nouvelle fois les trois gagnants : Elisabeth Hellec, Céline Chantry-Darmon et Cyprien Guérin.
N’hésitez pas à partager l’information autour de vous et à faire des commandes groupées dans vos labos. On compte sur vous pour nous donner vos impressions et partager vos objets/visuels favoris sur les réseaux (X ; BlueSky ; LinkedIn).